こんにちは! データサイエンス室の山本(@__Y4M4MOTO__)です。
ヤプリではdbtを用いてデータ基盤を運用しており、dbt docsをデータカタログとして使用しています。
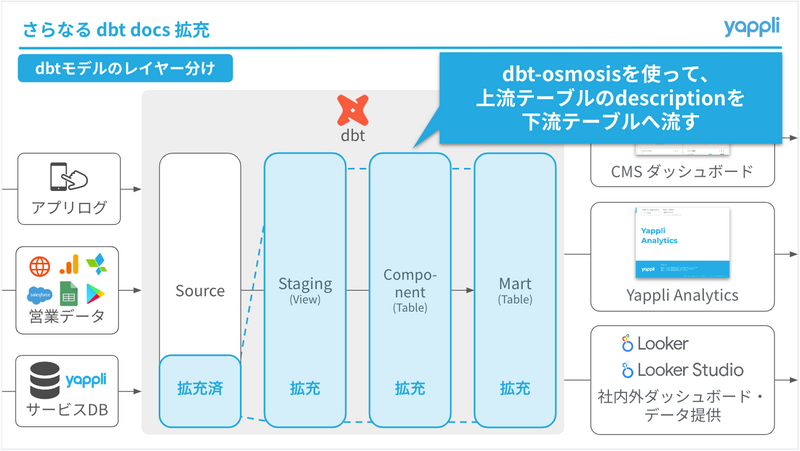
ヤプリのdbt基盤には主にアプリユーザーの行動ログやアプリプラットフォーム「Yappli」のサービスDBのデータが入っています。これらのデータをSource, Staging, Component, Martの4つの層で加工しています。当時、dbt docsのdescriptionが埋まっていたのはSource層のテーブルのみで他の層はほとんど埋まっておらず、拡充率としては30% ほどでした。
そこで、dbt-osmosisというツールを導入して拡充率を80% まで引き上げました。今回は、その導入過程と、その結果についてお話しします。
dbt-osmosisとは?
公式リポジトリはこちら。
dbt-osmosisはYAMLファイル(ソースYAMLファイル、スキーマYAMLファイル)を自動生成したり、上流テーブルのカラムのdescriptionなどを下流へ伝搬させたりするツールです。
これにより、同じカラムであれば上流テーブルのカラムのdescriptionさえ記載・更新すれば、下流テーブルのカラムのdescriptionも自動で更新されるため、効率的にメタデータを管理できます。
dbt-osmosisの有用性は次のスライドのp19~21がわかりやすいです。
dbt-osmosisの具体的な使い方については次の記事にまとめていますので、そちらをご覧ください。
導入方針
導入にあたって悩んだのが「dbt-osmosisの実行結果をGit管理するかどうか」です。
これを決めるにあたって、管理した場合としなかった場合でそれぞれどのような運用になるのかを考え、それぞれメリット・デメリットを洗い出しました。
はじめに: 当時のdbt docsの運用フロー
メリット・デメリットについて述べる前に、当時のdbt docsの運用フローについて簡単に説明します。
ヤプリのdbt基盤では主に次の3種類のブランチを使用しています。
- topicブランチ ... 新規開発・既存改修を行なうブランチ。チケットごとに作られる
- developブランチ ... 開発の主軸となるブランチ
- mainブランチ ... 本番環境のコードを置いておくブランチ
これらのブランチは次のようなフローで運用されています。
(新規開発・既存改修) topic-)develop: Pull Request develop-)main: Pull Request
(本番リリース)
本番リリースのPRがmergeされたタイミングで、GitHub Actionsを用いてGitHub Pagesへdbt docsをデプロイするようにしています。
GitHub Actionsのワークフローでは次のような流れで処理を行なっています。
- mainブランチへのpushが行なわれたらワークフローを実行
- ワークフロー内にdbtの実行環境を作成
dbt docs generateコマンドを実行してdbt docs用のファイル(index.html,catalog.json,manifest.json,assets/*)を生成- 生成されたファイルをGitHub Pagesへデプロイ
Git管理する場合
メリット
✅ dbt-osmosisの実行結果をPRレビュー等でチェックできる
dbt-osmosisの実行結果をGit管理するということは、当然その内容をPRでチェックすることになります。これにより、dbt-osmosisの実行結果に誤りがあった場合にPRレビューで指摘できるため、早期に修正できます。
✅ 本番のdbt docsへの反映方法に変更を加えなくて済む
dbt-osmosisの実行結果はPRを通してすでに取り込まれているはずであるため、これまでどおり本番のdbt docsへ反映すればOKとなります。
デメリット
❌ 一度書き込まれたdescriptionを再伝搬させるのが困難
dbt-osmosisの伝搬はdescriptionが空の場合に行なわれます。dbt-osmosisの実行結果をGit管理する場合、初回実行以降はdescriptionが埋まった状態となります。そのため、既存のdescriptionを変更する必要があった場合にそのままでは再度伝搬させることができません。
こういった場合に対応できるよう、dbt-osmosisには --force-inheritance というオプションが用意されています。このオプションを指定すると、すでにdescriptionが埋まっていてもその内容を上書きするようになります。これにより再伝搬が可能となります。
ただし、同じカラム名でも微妙に定義などが異なり、その旨がdescriptionに追記されていた場合、それらは上書きによって消されてしまいます。その場合、次のように meta: に osmosis_keep_description: true という設定を追加することでdescriptionの上書きを抑制できます。
- name: first_name description: "ここだけ手動変更" data_type: INT64 meta: osmosis_keep_description: true
ただし、dbt-osmosisは meta: の内容も伝搬するため、 osmosis_keep_description: true も伝搬されます。そのため、 osmosis_keep_description: true を設定したカラム以降のカラムについては再伝搬ができなくなります。
Xのリプライにて補足をいただきました!(ありがとうございます…!🙏 )
meta:の伝搬については--skip-merge-metaオプションを利用することで伝搬させないようにできるようです。
product.10x.co.jp
❌ YAMLファイルの行数が多くなり、人間がメンテナンスするにはつらい
dbt-osmosisを実行すると全テーブル全カラムについて、次のような name: と description: の2項目がYAMLファイルへ書き込まれます。
- name: customer_id description: "hogehoge"
そのため、YAMLファイルの行数がかなり多くなります(例えば、10カラムあるテーブルが10個あったら2 × 10 × 10 = 200行になります)。今後、手動でdescriptionを追加・修正する際に、膨大な行の中から目的の行を探すのはかなりの負担になることが予想されます。実際にdbt-osmosis実行後のYAMLファイルを手動更新してみましたが、地味ながら想像以上の負担でした。
dbt-osmosisではdbtモデルごとにスキーマYAMLファイルを分割させることもできるので、dbt-osmosisの実行結果をGit管理したい場合はそうした方が良さそうです。ただし、ファイル数が2倍に増えるため、その点については考慮する必要がありそうです。
❌ dbt-osmosisの実行し、その結果を取り込むワークフローを組む必要がある
dbt-osmosisの実行結果を取り込むために、どこかのタイミングでdbt-osmosisを実行する必要があります。手動実行するのは運用面で現実的ではないのでGitHub Actionsなどを使って自動化することになります。
当時のdbt docsの運用フローを踏まえ、Git管理する場合は次の2か所のいずれかが自動化タイミングになると考えました。
- topicブランチ → developブランチへのPR作成時にdbt-osmosisを実行し、その結果をPRに含める
- topicブランチ → developブランチへのPRがマージされたときにdbt-osmosisを実行し、その結果を取り込むためのPRを作成する
Git管理しない
メリット
✅ YAMLファイルには手動入力した分の記載しかないため、人間がメンテナンスしやすい
YAMLファイルに記載されるのは新たに追加したカラムや同名だが定義が変わっていて要補足なカラムのdescriptionが主となるため、人間でもメンテナンス可能な行数に収まります。
✅ 既存の開発フローに取り入れやすい
基本的に本番のdbt docsを更新するワークフローにdbt-osmosisを実行するステップを追加するだけで済むため、dbt基盤の開発メンバーに対して開発フローの変更を強いることなく導入できます。
✅ descriptionの再伝搬が容易
dbt-osmosisの実行結果をGit管理しないことで、常にdbt-osmosisを初回実行している状態となります。これによりdescriptionの再伝搬も容易に実現できます。
デメリット
❌ 本番反映前にdbt-osmosisの実行結果についてチェックすることが困難
dbt-osmosisの実行結果をGit管理しないため、どこかでチェックするフェーズを設けない場合、dbt-osmosisの実行結果が確認できるのは本番のdbt docsへ反映された後となります。
本番前にチェックするには別途dbt docs確認用の環境を用意する必要があります。しかし、dbt docs確認用の環境を新たに用意するのは意外と手間がかかります。
dbt docsの確認環境を用意方法として次の3つの方針が考えられます。
- ① GCSやS3にHTTPSとしてホスティングする
- ② 別のGitHubリポジトリを用意し、そのリポジトリのGitHub Pagesにホスティングする
- ③ ローカルで確認できるようにする
当初は①の方針を検討していましたが、社内公開に限定するためのアクセス制限に必要となる工数が予想以上だったため、断念しました。
続いて ② の方針も検討しましたが、dbt docsが確認できるメリットより運用が複雑になるデメリットの方が大きいと判断し、こちらも断念しました。
結果、Git管理しない場合は ③ の方針で進めるのが良いと判断しました。
どう導入したのか?
「Git管理する/しない」でメリット・デメリットを比較した結果、「Git管理しない」ことに決めました。決め手になったのは次の点です。
- Git管理する場合のデメリット: ❌ 一度書き込まれたdescriptionを再伝搬させるのが困難
- Git管理しない場合のメリット: ✅ descriptionの再伝搬が容易
メタデータへの追記・訂正が容易に行なえることは、有用なデータカタログを目指す上で欠かせないと考えました。
- Git管理する場合のデメリット: ❌ YAMLファイルの行数が多くなり、人間がメンテナンスするにはつらい
- Git管理しない場合のメリット: ✅ YAMLファイルには手動入力した分の記載しかないため、人間がメンテナンスしやすい
メタデータの追加・更新は人間が行なうことになるため、人間にとってメンテナンスしやすいかどうかは重視したいと考えました。
以上の判断をもとにdbt-osmosisの導入を進め、最終的に次の2か所に取り入れました。
- 本番のdbt docs
- ローカルのdbt docs
本番のdbt docs
dbt docsをGitHub PagesへデプロイするためのGitHub Actionsワークフローの流れを再掲します。
- mainブランチへのpushが行われたらワークフローを実行
- ワークフロー内にdbtの実行環境を作成
dbt docs generateコマンドを実行してdbt docs用のファイル(index.html,catalog.json,manifest.jsonassets/*)を生成- 生成されたファイルをGitHub Pagesへデプロイ
ここの手順3. のところでdbt-osmosisを実行するようにしています。ワークフローファイルから該当ステップを抜粋したものを次に示します。
- name: Generate dbt document run: | source venv/bin/activate dbt-osmosis yaml refactor --target prod --skip-add-data-types --add-progenitor-to-meta dbt docs generate
dbt-osmosisを実行してYAMLファイルを更新した後に dbt docs generate コマンドを実行することで、カラム情報が伝搬した後のメタデータでdbt docsが生成されます。
dbt-osmosisの実行は dbt-osmosis yaml refactor に次の3つのオプションをつけて行なっています。
--target prod--skip-add-data-types--add-progenitor-to-meta
--target prod は本番用のdbtプロファイルを使用する(=本番テーブルのメタデータを使用する)ことを意味しています。dbt-osmosisではローカルのYAMLファイルだけでなくデータウェアハウス上(ヤプリの場合BigQuery上)のテーブルのメタデータも使用します。今回は本番のdbt docsを更新したいため、本番用のプロファイルを使用するよう指定しています。
--skip-add-data-types はYAMLファイルに data_type: INT64 のようなデータ型を追記しないことを意味しています。データ型はYAMLファイルに記載しておかなくてもdbt docsに載るため、処理をスキップする目的で指定しています。
--add-progenitor-to-meta はカラムの伝搬元となるモデルを追記することを意味しています。これにより、カラムのdescriptionを追加・修正する必要があった際にどのモデルのカラムに対して行なえば良いかがわかります。 伝搬元は次のように meta: の osmosis_progenitor: という項目に記載されます。
- name: customer_id description: '' meta: osmosis_progenitor: model.my_dbt_project.stg_customers
ローカルのdbt docs
ローカルでもdbt-osmosis実行後のdbt docsを確認できるようにしました。これにより、 開発段階でdbt-osmosisの実行結果をチェックでき、前述のデメリットを解消しました。
確認は次の手順で行なえるようにしました。
- YAMLファイルに変更を加える
- 変更をコミットする
make docs-generateというコマンドを実行する
あらかじめ変更をコミットしておく必要がありますが、 dbt docs generate コマンドと似たような感覚で使えるようにしました。
手順3. ではMakefileに定義したこれらのコマンドを実行しています。
.PHONY: docs-generate
docs-generate:
# 後で戻って来れるようカレントブランチを取得
$(eval CURRENT_BRANCH=$(shell git branch --show-current))
# dbt-osmosis を実行するためのブランチを新規作成して checkout
git checkout -b review-dbt-docs
# カレントブランチの変更内容を review-dbt-docs へ取り込み
git merge --no-commit $(CURRENT_BRANCH)
git commit --allow-empty -m "Merge $(CURRENT_BRANCH) into review-dbt-docs"
# dbt-osmosis でカラム情報を流して artifacts を更新
dbt-osmosis yaml refactor --target prod --skip-add-data-types --add-progenitor-to-meta
dbt docs generate --target prod
# 役目を終えた review-dbt-docs ブランチを処分して元のブランチに戻る
git checkout .
git checkout $(CURRENT_BRANCH)
git branch -D review-dbt-docs
上記Makefileですが、下記の修正を行いました。
dbt-osmosis yaml refactor --target prod --skip-add-data-types --add-progenitor-to-meta - dbt docs generate + dbt docs generate --target prod理由は、 `dbt docs generate` でもtargetを指定しておかないと、デフォルトtargetを用いて(=dev環境のDBを参照して)dbt artifactsが生成されてしまうためです。
dbt-osmosisは既存のYAMLファイルに変更を加えます。dbt-osmosisが加えた変更はGit管理しないようにしたかったので、dbt-osmosisを実行するためのブランチを別途用意するようにしました。これにより、既存のYAMLファイルに変更を加えることなくdbt-osmosisの実行結果をdbt docs上で確認できるようになります。
ただし、この方法には次の注意点があります。
- コミット前に
make docs-generateコマンドを実行すると未コミットの変更を消し飛ばしてしまう可能性がある make docs-generateコマンド内でエラーが出るとdbt-osmosis実行用ブランチにとどまってしまうため、現状復帰に一手間かかる- 「一手間」=元のブランチに戻ってdbt-osmosis実行用ブランチを削除
この辺りは今後改善していければと思っています。
やってみた所感
dbt-osmosisで拡充できる範囲には限界がある
dbtモデルのカラムは上流モデルから流用されているものばかりではありません。途中で新しく追加したカラムや定義が変わったカラムなども存在します。そういったカラムのdescriptionは引き続き人力で埋める必要があります。
こちらについてはdbt-osmosisの公式ドキュメントでも触れられています。
今回の件でも、dbt-osmosis導入直後のdbt docsの拡充率は60%ほどで、そこから人力でカラムを埋めることで80%まで引き上げています。
osmosis_progenitor: に記載されている伝搬元が誤っている場合がある
Xのリプライにて補足をいただきました!(ありがとうございます…!🙏 )
こちら、不具合ではなく仕様のようです。
dbt-osmosisは伝搬元をクエリを解析して決定しているわけではなく、ノードの距離で決める仕様とのことです。そのため、ノードの距離が同一の場合は正確に判断できなくなります。
github.com
どのような場合に誤ってしまうのかはまだ検証中ですが、誤ってしまう場合の一例を以下に示します。
次のようなリネージグラフのモデル群があるとします(fct_item_shops のところの A ~ C はJOINする順でアルファベットを割り当てています)。
USING (shop_id) stg_items --> fct_item_shops: LEFT JOIN items C
USING (item_code) stg_item_shops --> fct_item_shops: shop_items A
実際のコードは次のリポジトリにあるのでそちらをご覧ください。
この場合、 fct_item_shops の各カラムの伝搬元はそれぞれ次のようになっているはずです。
item_key...raw_itemsitem_code...raw_shop_itemsshop_id...raw_shop_items
しかし、実際の結果は次のようになっており、 item_code の伝搬元が raw_shop_items ではなく raw_items となっています。
item_key...raw_itemsitem_code...raw_items←raw_shop_itemsのはずでは…?shop_id...raw_shop_items
dbt-osmosisの実行結果
version: 2 models: - name: stg_shops columns: - name: shop_id description: '' data_type: INT64 meta: osmosis_progenitor: source.my_dbt_project.tmp_dbt_osmosis_test.raw_shops - name: item_key description: '' data_type: STRING meta: osmosis_progenitor: source.my_dbt_project.tmp_dbt_osmosis_test.raw_shops - name: stg_item_shops columns: - name: item_code description: '' data_type: STRING meta: osmosis_progenitor: source.my_dbt_project.tmp_dbt_osmosis_test.raw_item_shops - name: shop_id description: '' data_type: INT64 meta: osmosis_progenitor: source.my_dbt_project.tmp_dbt_osmosis_test.raw_item_shops - name: stg_items columns: - name: item_key description: '' data_type: STRING meta: osmosis_progenitor: source.my_dbt_project.tmp_dbt_osmosis_test.raw_items - name: item_code description: '' data_type: STRING meta: osmosis_progenitor: source.my_dbt_project.tmp_dbt_osmosis_test.raw_items - name: fct_item_shops columns: - name: item_key description: '' meta: osmosis_progenitor: source.my_dbt_project.tmp_dbt_osmosis_test.raw_items data_type: STRING - name: item_code description: '' meta: osmosis_progenitor: source.my_dbt_project.tmp_dbt_osmosis_test.raw_items data_type: STRING - name: shop_id description: '' meta: osmosis_progenitor: source.my_dbt_project.tmp_dbt_osmosis_test.raw_shops data_type: INT64
暫定対応として、descriptionに正しい伝搬元を手動で記載しています。
この現象についてはdbt-osmosis公式リポジトリにissueとして報告しています。原因と解決策がわかったらPull Requestを作成したいと思います。
結び
この記事ではヤプリのdbt基盤にdbt-osmosisを導入した話について記しました。この記事がdbt-osmosis導入の参考になれば幸いです。
ここまでお読みいただきありがとうございました!この記事を読んでヤプリのデータ基盤開発に興味が出た方、ぜひカジュアル面談でお話ししましょう!